A multimodal whole-slide foundation model for pathology

作者:
Tong Ding1,2,3,4,19, Sophia J. Wagner 1,5,6,19, Andrew H. Song 1,2,3,19,
Richard J. Chen
期刊:nature medicine
重要指数:⭐️⭐️⭐️⭐️⭐️
时间:25.8.27
关键词:全切片基础模型\视觉–语言对齐\ALiBi位置编码
摘要:计算病理学领域因基础模型的最新进展而发生变革,这些模型通过自监督学习将组织病理学感兴趣区域(ROIs)编码为通用且可迁移的特征表示。然而,将这些进展转化为解决患者和切片层面复杂临床挑战的方法仍受限于特定疾病队列中临床数据的不足,尤其是针对罕见临床病症。我们提出基于Transformer的病理图像与文本对齐网络(titan),这是一个多模态全切片基础模型,通过视觉自监督学习和视觉-语言对齐技术,结合对应病理报告及423,122条由多模态生成式AI病理助手生成的合成标题,使用335,645张全切片图像进行预训练。该模型无需任何微调或临床标签,即可提取通用切片表征并生成可推广至资源有限临床场景(如罕见病检索和癌症预后)的病理报告。我们在多种临床任务中评估了titan,发现其在包括线性探测、少样本与零样本分类、罕见癌症检索、跨模态检索及病理报告生成在内的机器学习场景中,均优于ROI和切片基础模型。
1.研究背景
-
过去 3 年,病理 AI 的“基础模型”几乎全是 patch-level(256×256 像素小图)的自监督模型。它们把局部组织块压缩成 768 维向量,再在上面接一个小分类器,就能做癌症分型、突变预测。临床真实需求却是 slide-level(整张 10 万×10 万像素)任务:一张切片可能包含肿瘤、间质、坏死、正常腺体等多种区域,patch 模型只能“盲人摸象”。罕见病、小样本场景下,重新训练整张切片的模型代价极高。
-
目前已有 slide 模型,存在很多不足:只考虑视觉单模态、数据小泛化不足、必须针对每个新任务微调,不能像CLIP那样用文本提示直接分类检索
2.研究框架
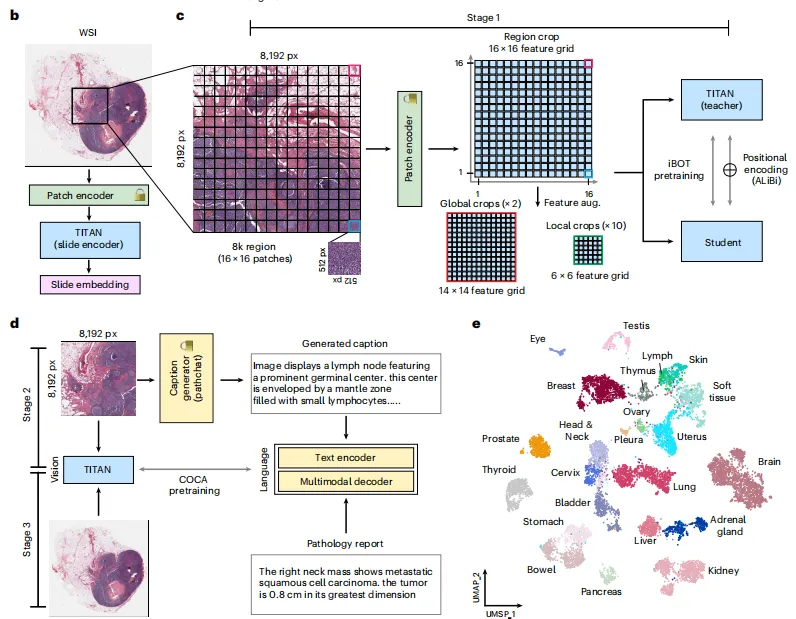
Patch encoder:使用 CLAM 工具箱对 WSIs 进行组织分割:在 HSV 低分辨率饱和度通道二值化后,经中值模糊、形态学闭合及面积滤波,提取 20× 下 512×512 像素非重叠 patch。特征由 CONCHv1.5(CoCa 目标、126 万图文对、20 epoch)提取,支持 FFPE、冷冻、IHC 等多染色类型,鲁棒且将序列长度缩短 4 倍,构成 2D 特征网格,供后续三阶段使用。
预训练protocol:iBOT 是一种把学生-教师知识蒸馏与掩码图像建模结合起来的自监督方法。
因为我们直接在 patch 特征空间 而不是原始图像上训练,所以做了以下调整:
视图生成
从一张 WSI 里随机切出 8 192×8 192 像素的 ROI(对应 16×16 的 512×512 patch 特征网格)。
每个 ROI 再切成:
– 2 个全局视图(14×14 网格)
– 10 个局部视图(6×6 网格)
这些视图不缩放、不插值,直接送进 iBOT。
随机采样 ROI 相当于额外数据增强,每个 epoch 都能让模型看到同一张切片的不同区域。
特征空间增广
传统方法先在像素空间做颜色/几何增强,再提特征,计算量大且视角有限。
我们改用 冻结特征增广:在已经训练好的 ViT 特征空间里直接对特征做扰动,既省显存又保持多样性,已被证明在小样本任务中有效。
Titan使用ViT将 WSI 编码为幻灯片嵌入,在stage 1是仅基于视觉的单模态预训练,对ROI裁剪图像进行预训练。
在stage 2为ROI级生成形态学描述的跨模态对齐。PathChat是一款专为pathology88设计的先进多模态大型语言模型。为突破传统临床报告仅关注最终诊断的局限,我们引导PathChat生成感兴趣区域(ROI)的详细形态学描述,为模型捕捉复杂病理特征提供了重要训练数据,为不同ROI(8,192×8,192像素)生成了合成标题。PathChat 无法直接处理 8 k×8 k 的 ROI,因此先把 ROI 切成 64 个 1 k×1 k 的 patch,再用 K-means(K=16)聚类,每类抽 1 个 patch,共 16 个代表性 patch 送 PathChat 生成描述;最后用 Qwen2-7B-Instruct 重写,提升语言多样性。
CoCa:对比查询学全局向量,128 个重建查询学生成。
8×A100,batch 3 k,冻视觉背bone,只训语言模块。
第三阶段为 WSI 级跨模态对齐,整合了来自GTEx联盟129的182,862组幻灯片-报告数据集(来源于内部临床报告和病理记录),报告以患者为单位,信息混杂,故用本地 Qwen2-7B-Instruct 提取单张幻灯片描述,并剔除敏感/无关内容(如大体描述、医院及医生姓名、患者临床病史等)。此外,我们还运用与合成标题相同的改写策略来丰富报告文本。
随机裁 32 k×32 k 区域(64×64 patch)→ 同样 CoCa。
- 8×A100,batch 256,lr 更小,防视觉权重崩溃。
数据类型:预训练(第一阶段)的Mass-340K组织样本分布。
包含20个器官的335,645个全扫描图像(WSI),样本类型包括:苏木精-伊红(H&E)染色(89.7%)、 IHC(7.9%)、特殊染色(2.3%)及其他(0.1%);或由肿瘤组织(70.0%)、组织损伤反应(8.4%)、正常组织(4.7%)、炎症组织(3.4%)及其他(13.5%)组成的混合样本,扫描设备类型多样。Titan预训练(第二、三阶段)采用Mass-340K的子集样本,并附带配对的图注和医学报告。
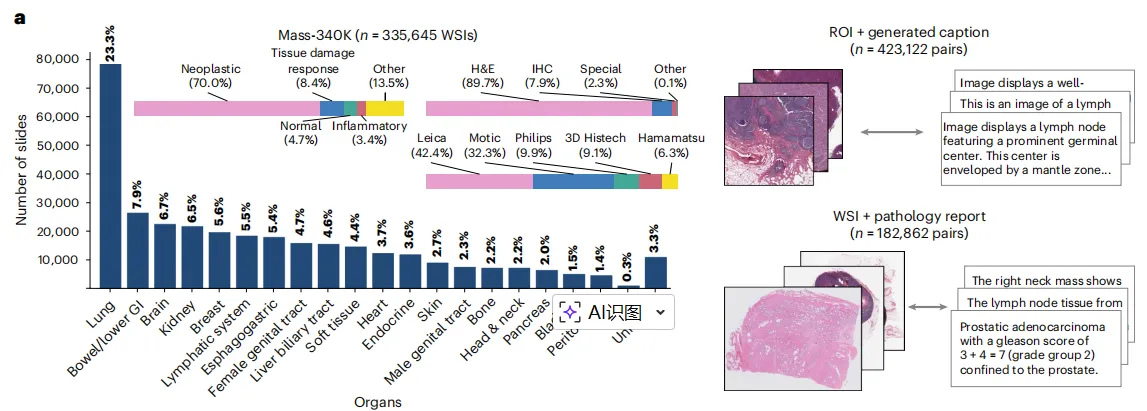
Fig 2 TITAN模型的性能
通过fig 2可得出,TITAN-V(红)、PRISM(绿)、GigaPath(黄)、CHIEF(蓝)
a) 数据量 vs 准确率,数据越多,性能单调上升;TITAN 数据效率更高。;
b) 参数量 vs 准确率,TITAN 用 一半参数 达到更高性能,说明架构效率。;
c) 线性探针评估Titan与基线模型在形态学分类、分子状态和生存预测任务中的表现。均值池化基线模型采用与Titan相同的补丁编码器(CONCHv1.5)。14 项形态学、39 项分子、6 项生存任务等对比,所有任务 P<0.0001(****);
d) 位置编码/Transformer层数/视觉预训练阶段的消融,ALiBi、6 层、8 k 区域最优;
e) 不同学习范式下滑片编码器性能变化的平均值,自监督特征质量高,微调收益大。;f) Few-shot 学习曲线:线性探测 > ABMIL;1-shot 场景 TITAN 领先 20%+。即使每类只有 1 张切片,TITAN 也能比传统监督方法准一半以上,且数据越少优势越大。
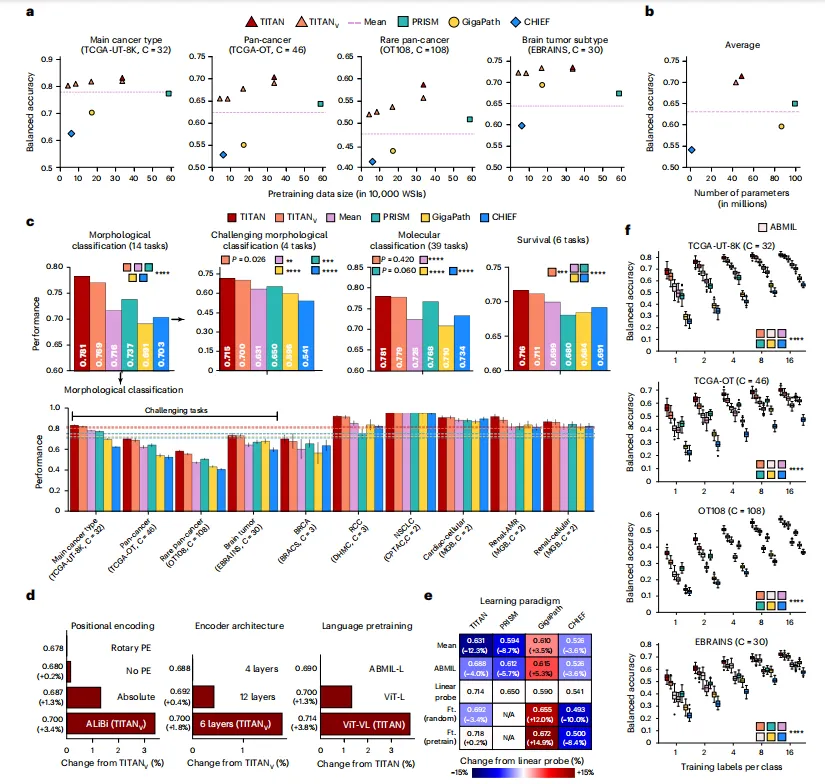
Fig 3 Titan的视觉语言评估
3a | 零样本评估示意图。通过识别幻灯片嵌入空间中最接近的文本提示嵌入来对查询幻灯片进行分类。
-
输入:WSI → slide embedding。
-
文本提示:“An example of {class}”。
-
输出:cosine similarity 最高的类别。
3b | Titan与Prism的零样本性能。
-
13 项任务(x 轴),balanced accuracy/AUROC(y 轴)。
-
数字:
-
平均多类任务:TITAN 0.714 vs PRISM 0.456(+56.5 %)。
-
EBRAINS(30 类):TITAN 0.598 vs PRISM 0.270(+121.9 %)。
-
-
结论:图文对齐带来真正的零样本能力。
-
为理解不同设计考量如何影响零样本性能,我们对预训练阶段和幻灯片编码器架构进行了消融实验(图c)
3c | 消融研究比较不同预训练策略
-
四组:共实验了四种Titan变体,展示了其在四个具有挑战性的亚型任务(TCGA -UT-8K、 TCGA -OT、OT108和ebrains)中的平均性能。仅 Stage 1(−7.3 %)、仅 Stage 2(−3.6 %)、仅 Stage 3(−0.4 %)、完整 TITAN。
-
结论:三阶段缺一不可,Stage 3(WSI-报告对齐)贡献最大。
3d | TCGA -SlideReports上的报告生成评估
-
指标:METEOR、ROUGE-1、BLEU-1。
-
数字:TITAN 比 PRISM 平均 +161 %(METEOR 0.35 vs 0.13)。
-
统计:P<0.0001。
3e | Titan与Prism生成报告的 TCGA 示例及对应临床报告。
-
三列:真实报告、TITAN 生成、PRISM 生成。
-
关键差异:TITAN 正确描述“Gleason 4+5=9、LVI、神经侵犯”,PRISM 漏掉。
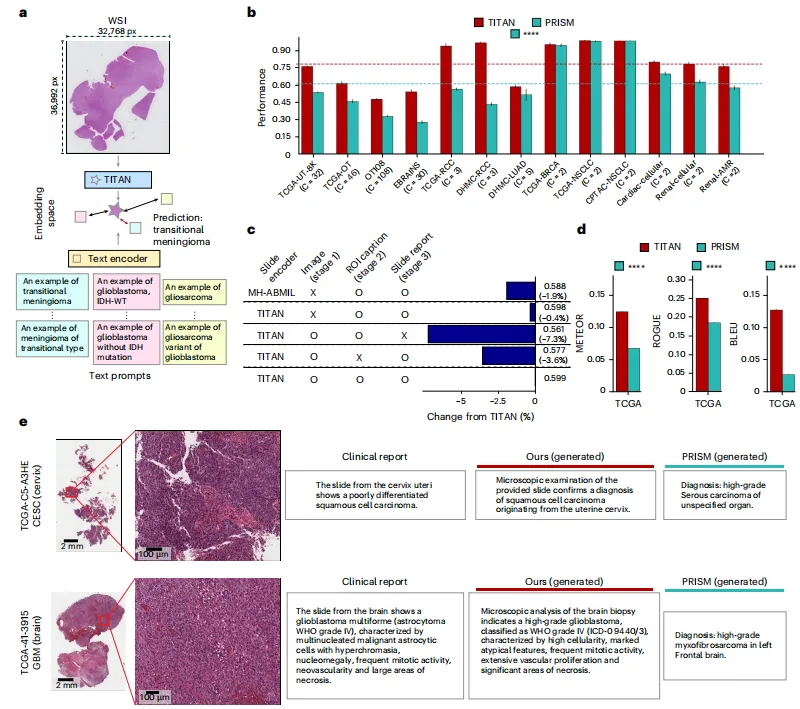
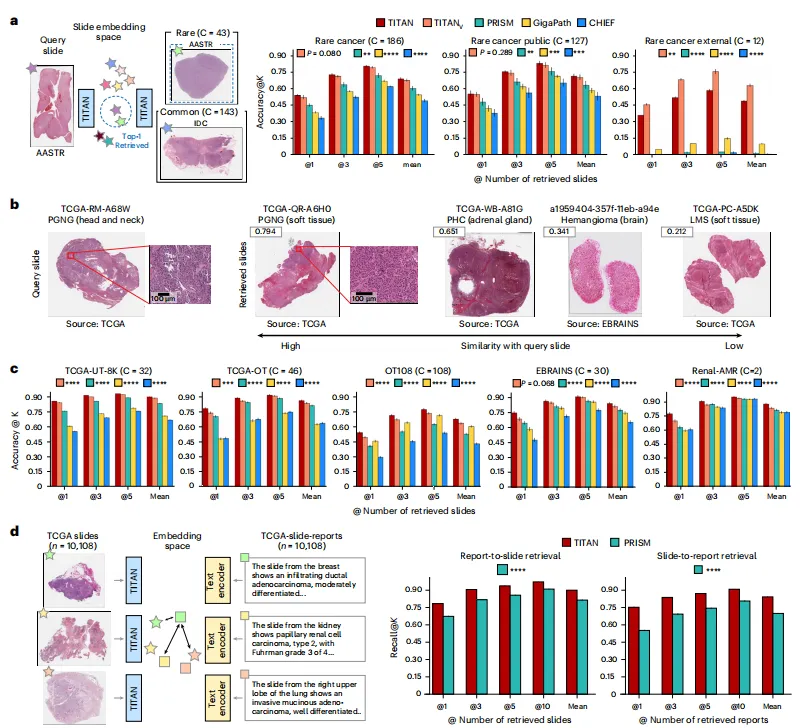
Fig 4 Titan的平台检索能力分析
4a | 基于Rare-Cancer(内部罕见癌症队列)的罕见癌症检索任务检索结果
-
三个任务:Rare-Cancer(186 类)、Rare-Cancer-Public(127 类)、Rare-Cancer-External(12 类)。
-
指标:Accuracy@1/3/5、MVAcc@5。
-
数字:
-
Rare-Cancer:TITAN Accuracy@5 = 0.90,PRISM 0.75(+14.8 %)。
-
External:TITAN Accuracy@5 = 0.85,GigaPath 0.54(+30.8 %)。
-
-
统计:所有 K 下 P<0.0001。
4b | Rare-Cancer队列中罕见癌症检索示例(查询切片及4个代表性检索切片)
-
查询:副神经节瘤(PGNG)。
-
Top-1:PGNG(相似度 0.794)。
-
Top-3:嗜铬细胞瘤(0.651)——临床相关。
-
结论:嵌入空间捕获了形态学相似性。
4c | 五项亚型分类任务的切片检索结果
-
均值代表三次实验的平均性能。五个任务(TCGA-UT-8K、TCGA-OT、OT108、EBRAINS、Renal-AMR)。
-
数字:TITAN 平均 Accuracy@5 0.88,PRISM 0.72(+16 %)。
4d | 基于Recall@K评估的跨模态检索性能(报告-切片与切片-报告)
-
Recall@K(K=1/3/5/10)。
-
数字:
-
Slide→Report:TITAN 平均 Recall@10 0.75,PRISM 0.55(+20.5 %)。
-
Report→Slide:TITAN 0.68,PRISM 0.58(+10.5 %)。
-
-
结论:图文空间真正对齐,支持“以图搜文”和“以文搜图”。
结果与讨论
- 做什么:我们提出 TITAN,一个基于 Transformer 的多模态全切片基础模型,把 patch 级的自监督学习、知识蒸馏和图文对齐成功扩展到整张切片。它在 14 项形态学、39 项分子和 6 项生存任务上全面优于现有 SOTA,尤其在罕见癌检索和零样本诊断场景下表现突出。
-
三阶段训练(视觉自监督 → ROI 图文对齐 → WSI 图文对齐)= 通用切片表征 + 零样本能力。
-
ALiBi 位置编码让“训练短、测试长”成为可能:16×16 小窗口训练,推理时直接外推到任意大切片,显存不爆。
- 为什么好:
-
数据规模 + 合成标题 + 真实报告 = 罕见癌也能学 33.6 万 WSIs + 42.3 万 ROI 描述 + 18.3 万报告,弥补罕见病数据缺口。
-
图文对齐是“多尺度信息”的关键 ROI 级描述学形态细节,WSI 级报告学诊断语义,二者缺一不可。
-
零样本 / 少样本性能碾压现有模型 1-shot 场景下,TITAN 比 ABMIL 高 56.7%,比 CHIEF 高 22.4%。
- 不足之处
①基于8k×8k区域裁剪数据进行预训练,并通过ALiBi外推至整个 WSI ,可能仍无法完整捕捉上下文信息。采用其他位置编码进行外推或许能解决这一局限。
②尽管我们竭尽全力构建了多样化的预训练数据集,但补丁基础模型及由此衍生的切片基础模型仍易编码非生物学特征(如组织处理场所和扫描仪),这可能影响其转化应用价值。我们认为,通过开展类似我们稳健性分析的系统性研究124,125,并持续构建更大规模的多机构预训练数据集,可有效缓解该问题。
③视觉-语言对齐技术处理临床报告仍是当前面临的挑战。整合有助于对比学习的全面临床信息,同时确保其与形态学存在一定关联,即使采用自动化处理流程仍需大量人工调校。将报告重构为独特的形态学和分子特征,可能促进更有效的学习。
④与用于补丁编码器和幻灯片编码器的其他预训练数据集相比,Mass-340K包含的幻灯片数量较少。我们认为,Titan已具备的强劲性能,结合Mass-340K的扩展工作,将进一步提升性能。
⑤改进的补丁表征质量可能增强下游幻灯片编码器的质量。
总结
从数据规模、模型架构、训练策略到下游任务,所有图都用“数字+统计显著性”证明:TITAN 在 更少参数、更少数据、零样本/少样本/罕见癌/跨模态 场景下,全面且显著地优于现有 SOTA,并首次在病理领域实现了 “图文双向检索 + 高质量报告生成” 的临床可用能力。